Le code génétique naturel de toutes les formes de vie sur notre planète n'est reconnu que sous la forme de polymères d'ADN et d'ARN.
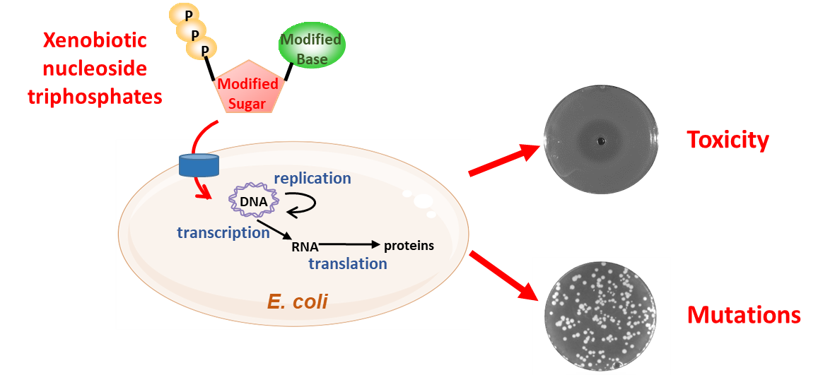
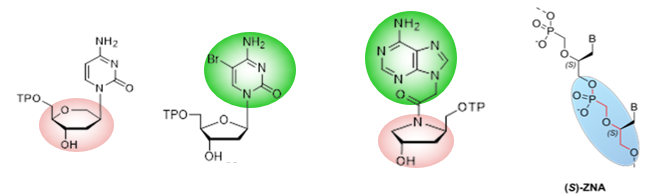
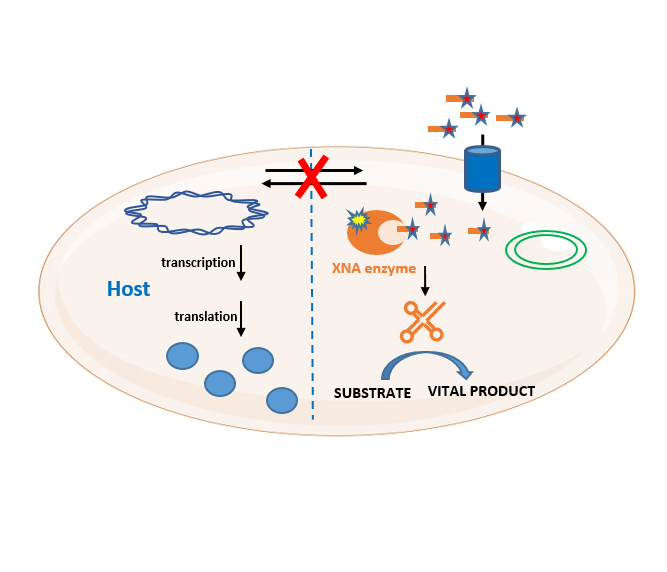
Une approche solide pour générer des OGMs sûrs consiste à développer un acide nucléique orthogonal, chimiquement distinct de l'ADN et de l'ARN, mais qui peut héberger des informations structurelles et/ou séquentielles essentielles à la viabilité et au phénotype des cellules. Un tel acide nucléique xénobiotique (XNA) sera génétiquement inerte et ne pourra être pris en charge que par des enzymes artificielles et synthétisé à partir de ses précurseurs xénonucléotides (XN).
Ces OGM « sûrs » ouvrent de nouveaux horizons pour les applications biotechnologiques et industrielles ; en tant que formes de vie non naturelles, ils contribuent également à une meilleure compréhension des organismes naturels.
XNAs dans les systèmes artificiels
Notre objectif est de développer un système d'information artificiel basé sur un nouveau type d'acides nucléiques qui pourrait fonctionner in vivo indépendamment du système d'information naturel. Pour atteindre cet objectif, quatre étapes doivent être élaborées : la sélection de la chimie du squelette, le développement d'un système de transport dans E. coli, une évolution et adaptation des polymérases et la sélection des produits vitaux.
L'équipe Xenome conçoit, construit et fait évoluer des micro-organismes dotés de systèmes génétiques artificiels, dont l'information est portée par les XNA. La prolifération et l'activité de ces organismes sont conditionnées à l'apport de XNAs, les plaçant sous le contrôle strict de l'utilisateur.
The Xenome team designs, builds and evolves microorganisms with artificial genetic systems, whose information is carried by XNAs. The proliferation and activity of these organisms are conditioned to the supply of XNAs, placing them under the strict control of the user.
Diversification des acides nucléiques dans la nature
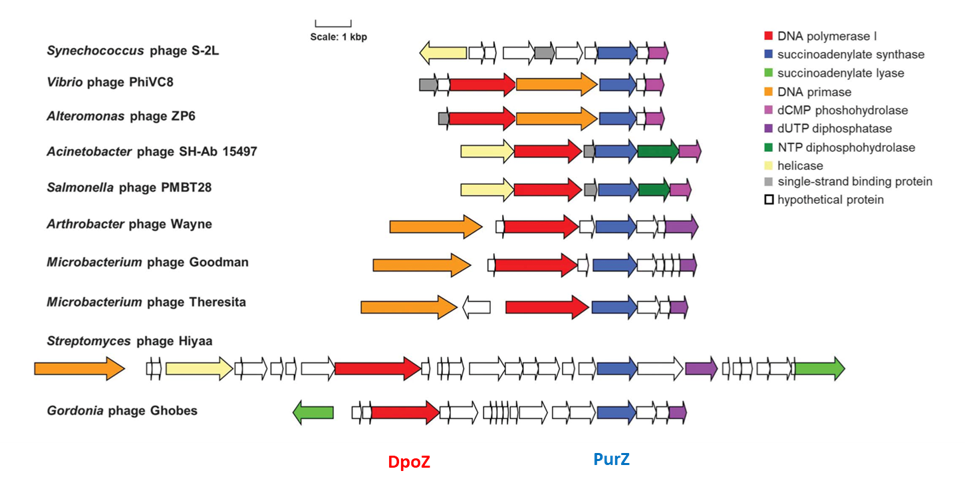
Nous étudions également la diversification des acides nucléiques dans le monde vivant naturel, en nous concentrant sur les génomes de bactériophages qui présentent une grande diversité chimique de bases nucléiques non canoniques. Certains virus infectant des hôtes tels que les protéobactéries, les cyanobactéries et les actinobactéries ont un génome à ADN dans lequel l'adénine est complètement remplacée par l'aminoadénine (Z). L'élucidation de la voie de biosynthèse de ce nucléotide non canonique et la découverte des ADN polymérases du bactériophage DpoZ dédiées à l'incorporation de ce nucléotide dans l'ADN ouvrent la voie à la propagation de génomes chimiquement modifiés chez les bactéries (Pezo et al., Science 2021).Pezo et al., Science 2021).
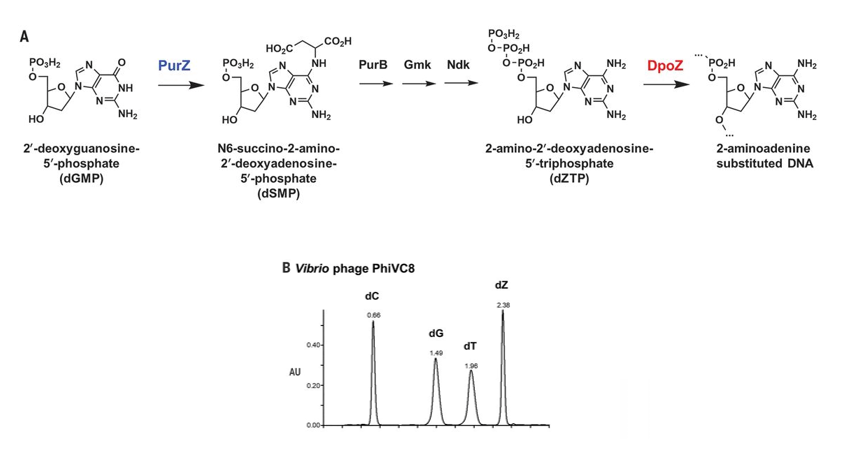
A _ Voie de synthèse du dZTP via des réactions catalysées par l'enzyme biosynthétique PurZ codée par le phage et l'ADN polymérase DpoZ. Les étapes marquées « PurB », « Gmk » et « Ndk » sont catalysées par des enzymes de l'hôte bactérien.
B _ Profil HPLC de l'ADN phagique digéré. AU, unités arbitraires ; dC, désoxycytidine ; dG, désoxyguanosine ; dT, désoxythymidine ; dZ, désoxyaminoadénosine ; dA, désoxyadénosine.
Figures extraites de Pezo et al, Science 2021
B _ HPLC profile of phage DNA digested by nuclease and phosphatase. AU, arbitrary units; dC, deoxycytidine; dG, deoxyguanosine; dT, deoxythymidine; dZ, deoxyaminoadenosine; dA, deoxyadenosine.
Figures extracted from Pezo et al., Science 2021